BrdUラベリングの時系列データからパラメータ推定
タグ: #research #R #parameter estimation #BrdU
はじめに
岩見真吾他(箸)のウイルス感染と常微分方程式 (シリーズ・現象を解明する数学)を元に時系列データから数理モデルのパラメータを推定する方法を解説する。
ウイルス感染と常微分方程式 (シリーズ・現象を解明する数学)
Amazon購入リンク:https://amzn.to/2JOWl5P
Rを使った最小二乗法によるデータフィッティングの方法に重点を置き、数理モデルの説明は最小限にする。
使用するデータは、岩見真吾, 佐藤佳, 竹内康博 (2017). 「ウイルス感染と常微分方程式」. 共立出版
詳細な導出は省くが、BrdU投与中、BrdU投与後の活性化T細胞BrdU陽性率は以下の式で表される。
Rを用いた最適化
Rの基本的な知識は
ライブラリの読み込みや標準の設定の準備
最初に使用する関数のライブラリを読み込むことと、計算に必要な基本的な設定をしておく。ここを編集すれば全体の計算の調整ができることが理想。
rm( list=ls(all=TRUE) )
library(ggplot2)
set.seed(0)
Tmin <- 0.0
Tmax <- 40.0
step_size <- 0.01
stime <- seq(Tmin,Tmax,step_size)
lbt <- 0.01 # lower bound threshold
ubt <- 100.0 # upper bound threshold
pars <- c(p = 1.0, d = 2.0, alpha = 0.5)
最初の行では前に定義したオブジェクトを消去する作業をしている。rm()は引数となるオブジェクトの定義を消去する関数。前の計算の結果を間違って使用しないようにするためのおまじない。
rm( list=ls(all=TRUE) )
使用する関数が定義されたライブラリを読み込む。今回は作図に必要なggplot2のみを読み込んでいる。
library(ggplot2)
乱数のシード値を設定する。set.seed()に渡す引数によってシード値が決定する。乱数は最適化関数などに使用される。
計算では擬似乱数(乱数のような数字の列)が用いられるので、シード値はどの数字の列を使うかを設定する。同じシード値を使って同じコードを書けば、同じ計算ができる。
set.seed(0)
モデルを計算する時間(カレンダータイム)を決めている。Tminが計算開始(実験開始)の時間、Tmaxが計算終わり(実験終了+α)の時間。step_sizeは時間の刻みはばを決める。コンピュータは連続時間を計算することができないので、離散化して計算することになる。stimeは計算する時間のベクトルでseq()でTminからTmaxまでの数をstep_sizeで刻んだ数列を保持している。
Tmin <- 0.0
Tmax <- 40.0
step_size <- 0.01
stime <- seq(Tmin,Tmax,step_size) # 0.00, 0.01, 0.02, 0.03,..., 39.98, 39.99, 40.00
lbtとubtはパラメータ探索の範囲を決める。後述。
lbt <- 0.01 # lower bound threshold
ubt <- 100.0 # upper bound threshold
parsにはパラメータの探索初期値が定義される。c()は引数をベクトルにする。p = 1.0というふうにかけば、各要素に名前がついたベクトルになる。
pars <- c(p = 1.0, d = 2.0, alpha = 0.5)
データの準備
#####################
### data
#####################
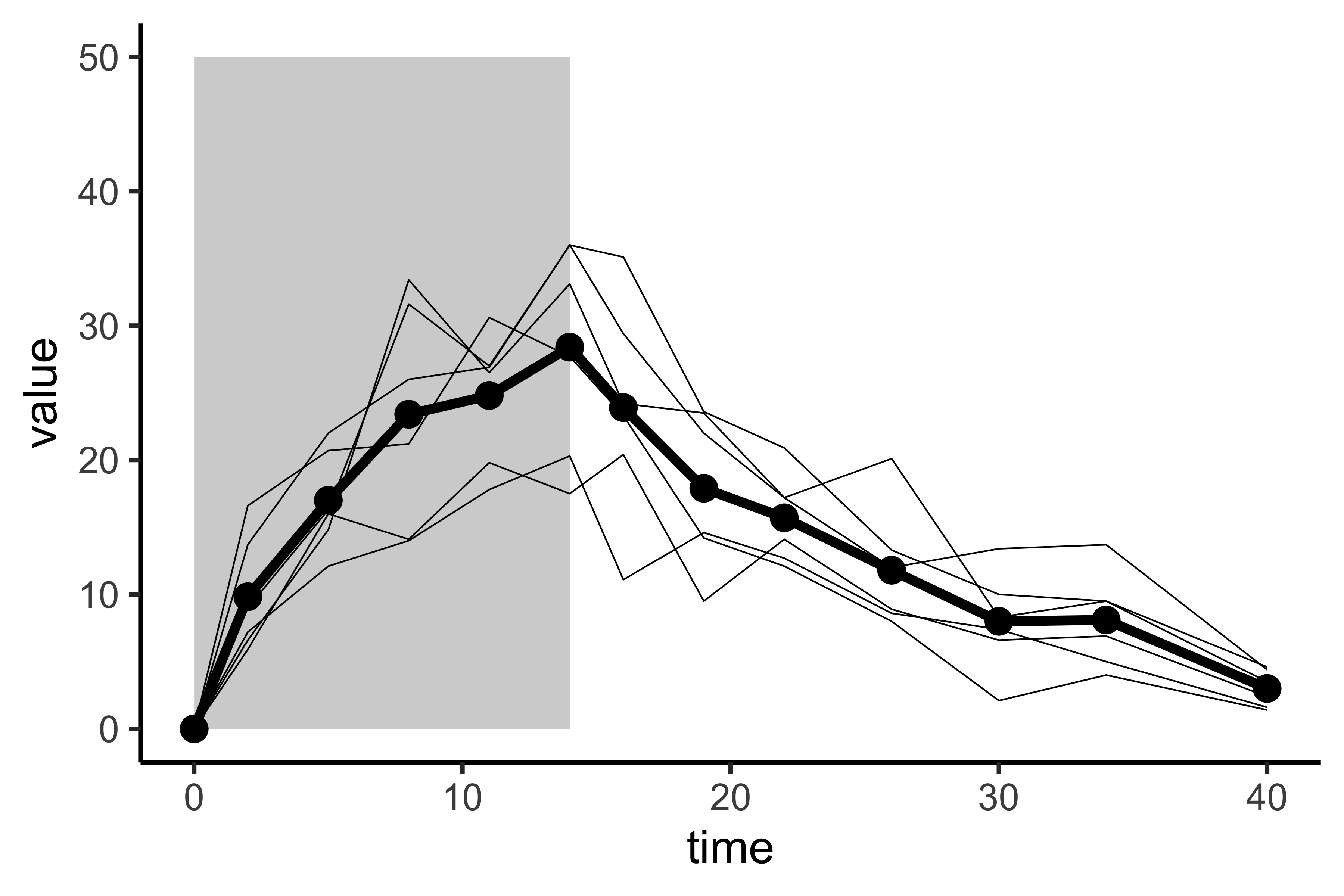
mouse_ids <- c("78F1","78F3","78F4","78F5","78M1","78M2")
expt_time <- c(0,2,5,8,11,14,16,19,22,26,30,34,40)
d78F1 <- c(0.00,7.2,12.1,14.0,17.8,20.3,11.1,14.6,12.7,8.6,7.4,5.0,1.6)
d78F3 <- c(0.00,6.6,14.8,33.4,26.5,33.1,24.2,23.5,17.2,12.0,13.4,13.7,4.4)
d78F4 <- c(0.00,9.0,16.3,31.6,27.0,36.0,29.4,22.0,17.2,20.1,8.3,9.5,4.6)
d78F5 <- c(0.00,13.7,22.0,26.0,26.9,36.0,35.1,23.6,20.9,13.3,10.0,9.5,3.5)
d78M1 <- c(0.00,5.9,16.0,14.1,19.8,17.5,20.4,9.5,14.1,8.9,6.6,6.9,2.4)
d78M2 <- c(0.00,16.6,20.7,21.2,30.6,27.7,23.3,14.2,12.1,8.0,2.1,4.0,1.4)
dmean <- c(0.00,9.83,17.0,23.4,24.8,28.4,23.9,17.9,15.7,11.8,8.00,8.10,3.00)
lowdata <- data.frame(time = rep(expt_time, length(mouse_ids)), value = c(d78F1,d78F3,d78F4,d78F5,d78M1,d78M2),id = rep(mouse_ids,each=length(expt_time)))
meandata <- data.frame(time = expt_time, value = dmean)
dbrdu <- data.frame(time=c(0,14),value=c(50,50))
data <- meandata
データファイルの準備の詳細は
Rでの時系列データの準備 | Shoya Iwanami’s note
データのプロット
まずは、データを図にしてみるところから始める。どのようなデータを解析するのか、どのような構造になっているのかを知ることができる。読み込んだデータと元のデータに間違いがないかどうかも確認できる。
ggplotの使い方は
ggolotの初歩(作成予定)
plt <- ggplot() +
geom_area(data=dbrdu,aes(x=time,y=value), fill="lightgray") +
geom_line(data = lowdata,aes(x=time,y=value,group=id),lwd=0.5,color="black") +
geom_line(data=meandata,aes(x=time,y=value),lwd=3,color="black") +
geom_point(data=meandata,aes(x=time,y=value),size=8) +
scale_y_continuous(limits = c(0,50)) +
theme_classic(base_size = 30,base_family = "Helvetica")
ggsave(filename = "./output/data_plot.png", plot = plt, width = 12, height = 8)